siRNA
Por siRNA se conocen a los pequeños RNA interferentes (del ingles small inference RNA). Este tipo de ARN bicatenario es de reciente descubrimiento, en 2006 se concedió el Nobel de Fisiología y Medicina a Craig C. Mello y Andrew Fire por el descubrimiento de la ruta que empleaban estos pequeños RNAs para interferir en la regulación génica. No obstante, una gran parte de su funcionamiento permanece todavía sin descubrir. Es por eso que, actualmente, los siRNAs se encuentran en la cresta de la ola de la investigación del silenciamiento génico.
Los siRNAs pueden ser de origen endógeno o más frecuentemente exógeno. La mayoría de los siRNAs estudiados tienen su origen en algún patógeno, principalmente virus, tanto de plantas y animales. Sin embargo pueden, en ocasiones, estar codificados por el propio organismo; por ejemplo en una secuencia repetida en tándem.
Independientemente de su origen, los siRNAs se forman a partir de un RNA de cadena sencilla que actúa como molde para una polimerasa dependiente de RNA, RDRPs (del inglés: RNA-dependent RNA polymerases). Estas polimerasas se unen a una molécula de RNA monocatenario, ssRNA (del ingés single stranded RNA) y sintetizan la hebra complementaria, formando un RNA de doble cadena, abreviado dsRNA (doble stranded RNA). Cabe recordar que existen virus de dsRNA que pueden actuar directamente sin necesidad de las RDRPs. De cualquier modo, a partir de un ARN de doble hebra de un tamaño relativamente largo se sintetizan varios siRNAs, aunque no todos los trozos resultantes del troceado son siRNAs funcionales. El dsRNA largo es cortado por una enzima llamada Dicer, una proteína citoplasmática de la familia de las RNAs III (una familia de enzimas cuya función es cortar RNA). Los trozos de dsRNA resultantes, ya siRNAs, tienen un tamaño comprendido entre 20 y 25 nucleótidos que tienen en el extremo 5″ un grupo fosfato y un grupo hidroxilo en el extremo 3″. La hebra negativa o anti-sense será la que se ensamble en el complejo proteico denominado RISC y que posteriormente se apareará por homología de bases con un mRNA. La homología entre el siRNA y el mRNA es muy alta, puesto que de los 25 nucleótidos tan solo hasta 4 pueden ser no homólogos.
El centro activo del gran complejo ribo-proteico RISC es a una proteína del tipo Argonauta (abreviada AGO en la literatura científica normalmente), con capacidad endonucleasa, que corta hebras de mRNA en puntos intermedios de la cadena. Los trozos resultantes pueden pasan a ser degradados por la maquinaria celular normal. Recientemente se ha propuesto que los trozos de mRNA cortados por AGO podrían entrar a formar parte del pool de siRNAs tras ser procesado por una nueva RDRP, distinta de la primera.
Gracias a los siRNA, se suprimen la expresión de genes mediante la degradación de sus mRNA. Además los siRNAs han sido descritos como participes de la organización de la cromatina. En la actualidad se están estudiando multitud de interacciones virus-hospedador puesto que se han descrito siRNAs en diversas rutas de defensa contra virus y como forma de ataque del virus. Durante el proceso de infección viral ambas partes procesan o sintetizan RNA para obtener siRNAs que silencien las defensas o bien las proteínas víricas.
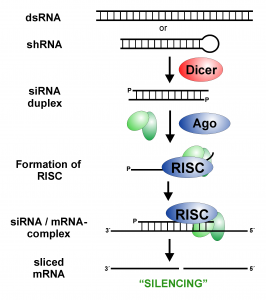
Esquema del silenciamiento génico producido por los siRNAs y el comlejo RISC
Su descubrimiento no solo ha permitido entender mejor la interacción virus-hospedador, sino que también ha resultado ser una excelente herramienta de laboratorio. El silenciamiento por siRNA ha resultado ser una excelente alternativa a los individuos mutados en su genoma para el estudio científico del funcionamiento de las proteínas. Permitiendo inhibir o disminuir la expresión de proteínas sin necesidad de mutagenizar. Cada día se describe alguna modulación de la expresión por siRNAs en procesos ya conocidos, dando una nueva dimensión a procesos que se creían muy bien estudiados.

