Splicing alternativo o barajado de exones
El splicing alternativo o barajado de exones es el mecanismo molecular de edición post-transcripcional mediante el cual se obtienen varios mARN (ARN mensajero, aquel que será traducido a proteínas) a partir de una única secuencia de ADN que ha sido transcrito a un transcrito primario de mARN. El fenómeno de splicng alternativo es fundamentalmente eucariota. Las bacterias y los virus pueden presentar este tipo de tratamiento para su ARN, pero es mucho menos frecuente. Mientras que las bacterias y los virus suelen tener genes solapantes para economizar su ADN los eucariotas han optado por el splicing, para dar con un número determinado de genes, muchas más proteínas, tirando por tierra el dogma de la biología de un gen una proteína. El splicing alternativo a pesar de ser preferentemente eucariota no es responsable de la complejidad de estos organismos, sino más bien es una consecuencia de ella.
El splicing alternativo hace referencia a las alternativas que tiene el ARN mediante el splicing, que es la eliminación de secuencias concretas de la cadena de ARN. De esta manera, con la reorganización o eliminación de un fragmento del ARNm, se pueden traducir proteínas con función o lugar de acción diferente, que serán llamadas isoformas de una misma proteína. Las isoformas conseguidas de esta u otra manera suelen diferenciarse por letras griegas tras el nombre.
En el ADN que codifica para proteínas encontramos dos tipos de secuencias, intrones y exones. Los primeros son secuencias intergéncias que no llegarán a ser traducidas a proteína, es decir, serán eliminados por splicing “convencional”, para saber más sobre los intrones o sobre los tipos de splicing que tiene cada tipo de intrón puedes leer el artículo que le dedicamos aquí. Los exones por otro lado serán las secuencias que sí se traducirán a proteínas, para saber más de ellos puedes ver el artículo que les dedicamos aquí.
Los exones no se reordenan al azar sino que el splicing está muy controlado a fin de que no surjan proteínas perjudiciales para el propio individuo.
Tipos de splicing alternativo:
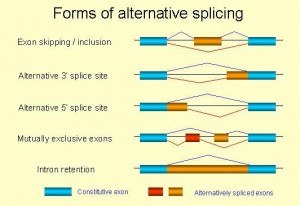
Formas de hacer splicing alternativo a) eliminación/inclusión de exones, b) 3″ alternativo (difenretes sitios de poliA) c) diferentes promotores y d) retención de intrones
El caso más sencillo es el splicing de exones, en el que un exón es eliminado junto con dos secuencias intrónicas colindantes.
Otra posibilidad es que un intrón no se elimine, y se traduzca a proteína, con la posibilidad de que codifique un codón de parada o cambie la pauta de lectura de la cadena de ARN.
Selección de promotores: un único gen está bajo la influencia de dos promotores (secuencias a las que se une la ARN polimerasa para formar el ARNm) y dependiendo de las circunstancias se activará un promotor u otro dando lugar a un extremo N-terminal diferente.
Selección de sitio de poliadenilación: la secuencia de ARNm tiene dos secuencias que señalizan la adición de una cola de varias adeninas, llamada cola poliA. Esta cola que presentan el 70% de los ARNm que se sirve para marcar el ARNm para su traducción a proteína y dependiendo de cual de las secuencias esté activada se producirá una proteína u otra.
El fenómeno de splicing alternativo también puede dar lugar a recombinación entre exones de diferentes genes, en este caso se denomina trans-splicing.
