Exones
Los exones de un gen son las secuencias del ADN que van a dar el ARN mensajero que será traducido a aminoácidos que darán la proteína que codifica ese gen (son las secuencias que se expresarán). Los exones están separados por intrones, regiones que no codifican para la proteína que son eliminados en el ARN mensajero antes de ser traducidos. Para saber más sobre los intrones puedes leer nuestro artículo sobre ellos aquí.
En 1978 el bioquímico Dr. Walter Gilbert definió a la luz de la necesidad de diferenciar cistrones de lo que tenían realmente. El exón quedó definido como la parte que llevaba la información que será traducida, sin embargo con el tiempo los intrones han resultado tener cierta utilidad.
Walter Gilbert llamó barajado de exones a su hipótesis de que estos renglones de información, los exones, podían ser barajados dando lugar o diferentes proteínas. Más tarde se observó este fenómeno y de forma moderna se le denomina splicing. Con este mecanismo de reorganización de los exones normalmente alguno de los exones es eliminado junto con los intrones. Por ejemplo la actina, una proteína muy importante para el citoesqueleto y la movilidad de las células presenta 6 genes y cada uno tiene 6 exones, que pueden dar lugar hasta 21 proteínas diferentes.
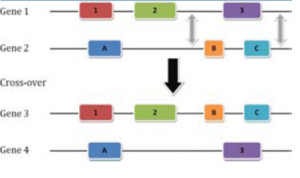
Segúl la teoría del barajado de exones surguian genes nuevos de mezclar exones de diferentes genes existentes.
Gilbert sugirió que cada exón codificaría para un domino proteico (aunque después se demostró que existen algunos dominios separados en más exones y algunos exones que contenían más de un dominio proteico). Este barajado puede dar lugar a nuevos genes por recombinación, sobrecruzamiento, exclusión o duplicación de exones. Puede ser que el exón asuma una nueva función después de barajarse, o que mantenga su función original. Debe ser un proceso muy antiguo, quizás originado en un mundo ARN primitivo. En la actualidad se calcula que el 19% de los genes de eucariotas se han formado así.
Además, es importante mencionar que el proceso de splicing no es aleatorio, sino que está altamente regulado y controlado por una serie de proteínas y moléculas de ARN pequeñas conocidas como snRNP (pequeñas ribonucleoproteínas nucleares). Estas moléculas reconocen las secuencias específicas en el ARN mensajero que indican dónde deben unirse los exones y dónde deben eliminarse los intrones.
Cuando se transcribe el ADN en ARN se forma un ARN mensajero que contiene tanto los exones como los intrones y estos últimos se eliminan durante la maduración del ARN mensajero, en este punto es en el que ocurre el splicing alternativo. Además otras moléculas de ARN como el ARNt o el ARNr (de transferencia y ribosómico respectivamente) también presentan procesos de maduración en los que se pueden eliminar ciertas secuencias a las que también se denominan intrones dejando los exones funcionales (que no se traducen a proteína).
Además de por splicing alternativo convencional en el que un único ARN pierde partes de su secuencia durante la maduración también puede ocurrir el fenómeno de trans-splicnig en el que los exones de dos genes diferentes se juntan para dar un ARN maduro, este proceso que ocurre de forma normal para algunas proteínas, también está asociado a proteínas mutantes en algunos cánceres.
La existencia de intrones y exones es una de las características de los genomas de eucariotas. Si bien es verdad que algunos genes de bacterias y arqueas presentan intrones, la gran mayoría contienen solo un transcrito que será traducido a proteína sin la maduración del ARNm que ocurre en eucariotas.
Es importante destacar que el estudio de los exones y su función en la codificación de proteínas ha sido fundamental para entender las enfermedades genéticas. Muchas de estas enfermedades son causadas por mutaciones en los exones que alteran la secuencia de aminoácidos de la proteína resultante, lo que puede afectar su función. Además, las técnicas de secuenciación del genoma han permitido identificar variantes de exones que están asociadas con ciertas enfermedades, lo que puede ayudar en el diagnóstico y tratamiento de estas condiciones.
